TIANScriptⅡ RT Kit
Vipengele
■ Shughuli kubwa ya enzyme na ufanisi: Shughuli kubwa ya transcriptase na utangamano mzuri katika majaribio yanayofuata.
■ Sehemu kubwa ya substrate: Inafaa kwa RNA zote, haswa templeti za RNA zilizo na miundo tata ya sekondari.
■ Urefu wa RT: Mchanganyiko wa kamba ya kwanza ya cDNA inaweza kufikia 12 kb.
■ Operesheni rahisi: Ongeza tu vitu vinavyohitajika kwa hatua moja bila kuongeza reagent yoyote wakati wa operesheni.
Maombi
■ Usanisi wa cDNA strand ya kwanza.
■ Ujenzi wa maktaba ya cDNA.
■ RT-PCR ya hatua moja.
■ Uchambuzi wa MBIO.
Bidhaa zote zinaweza kuboreshwa kwa ODM / OEM. Kwa maelezo,tafadhali bofya Huduma iliyoboreshwa (ODM / OEM)
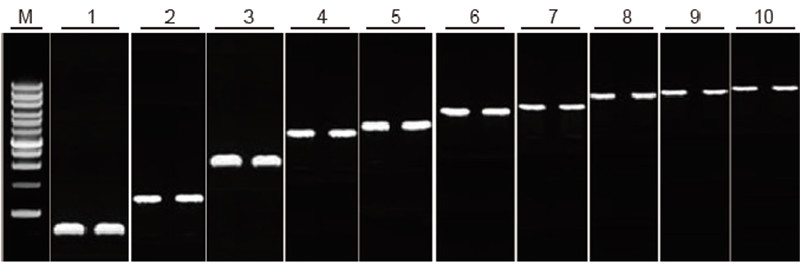 |
Badilisha uwezo wa ununuzi wa TIANScript II RT Kit kwa vipande tofauti vya urefu Njia: Rejea unukuzi: Rejea mwongozo wa maagizo ya TIANScript II RT Kit. Matokeo: Picha ya gel inaonyesha matokeo ya kukuza ya jeni za kulenga 10 zilizo na urefu tofauti baada ya nakala ya nyuma ya 1 μg ya jumla ya RNA. 2 μl ya bidhaa za nakala za nyuma zilipakiwa kwa kila njia. Mfumo wa kukuza (PCR): 20 μl; Mzigo wa mfano: 5 μl; Alama: D15000 + 1 kb Ngazi ya DNA; Mkusanyiko wa gel: 1%; Hali ya Electrophoresis: 6 V / cm, 20 min Mchoro wa kila mstari: M: Alama ya DNA; 1: Urefu wa bidhaa: 120 bp; 2: Urefu wa bidhaa: 1 kb; 3: Urefu wa bidhaa: 2.5 kb; 4: Urefu wa bidhaa: 3.2 kb; 5: Bidhaa urefu: 4.6 kb; 6: Urefu wa bidhaa: 6.8 kb; 7: Urefu wa bidhaa: 7.6 kb; 8: Urefu wa bidhaa: 8.9 kb; 9: Urefu wa bidhaa: 10 kb; 10: Urefu wa bidhaa: 12 kb; |
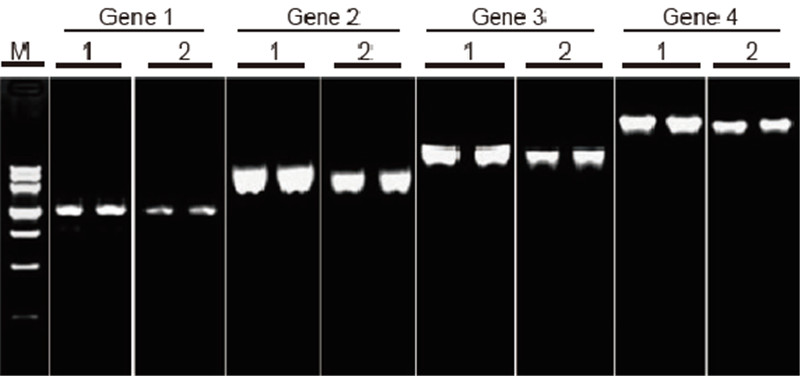 |
Kulinganisha ufanisi na maalum ya TIANScript II RT Kit na bidhaa kutoka kwa wasambazaji wengine kwa nakala ya nyuma ya templeti ndefu Vifaa: Jumla ya RNA ya seli zinazofuata binadamu. Kiasi cha kuanzia RT-PCR: 2 μl bidhaa ya kunakili ya nyuma (50 ng / μl) Njia: Rejea unukuzi: Rejea mwongozo wa maagizo ya TIANScript II RT Kit. Matokeo: Picha ya gel inaonyesha matokeo ya kukuza ya jeni za kulenga 6 zilizo na urefu tofauti baada ya nakala ya nyuma ya 1 μg jumla ya RNA ya seli zinazoshikilia binadamu kutumia M-MLV kutoka kwa Muuzaji A na TIANGEN TIANScript II RT Kit. Mfumo wa kukuza (PCR): 20 μl; Mzigo wa mfano: 5 μl; Alama: DNA AlamaIII; Mkusanyiko wa Gel: 1%; Hali ya Electrophoresis: 6 V / cm, 20 min. Mchoro wa kila mstari: M: Alama ya DNA; 1: Matokeo ya kukuza ya cDNA reverse transcribed kwa kutumia TIANScriptII RT Kit; 2. Matokeo ya kukuza ya cDNA iliyochapishwa kwa kutumia bidhaa inayofaa kutoka kwa Muuzaji A. Urefu wa bidhaa ya jeni 1 ni 1.3 kb; Urefu wa bidhaa ya jeni 2 ni 3.0kb; Urefu wa bidhaa ya jeni 3 ni 5.0 kb; Urefu wa bidhaa ya jeni 4 ni 7.5 kb. |
RNA-1 imeharibika
-Takasa RNA ya hali ya juu bila uchafuzi wowote. Nyenzo ambayo RNA hutolewa inapaswa kuwa safi iwezekanavyo ili kuzuia uharibifu wa RNA. Chambua uadilifu wa RNA kwenye gel iliyochorwa kabla ya majibu ya RT. Baada ya uchimbaji wa RNA, inapaswa kuhifadhiwa kwa 100% formamide. Ikiwa kizuizi cha RNase kinatumika, joto la joto linapaswa kuwa <45 ° C, na pH inapaswa kuwa chini ya 8.0, vinginevyo kizuizi kitatoa RNase yote iliyofungwa. Kwa kuongezea, kizuizi cha RNase kinapaswa kuongezwa katika suluhisho zilizo na ≥ 0.8 mM DTT.
A-2 RNA ina vizuizi vya athari za kurudia unukuzi
—— Vizuizi vya unukuzi mara kwa mara ni pamoja na SDS, EDTA, glycerol, pyrophosphate ya sodiamu, spermidine, formamide, chumvi ya guanidine, n.k Changanya RNA ya kudhibiti na sampuli, na ulinganishe mavuno na athari ya kudhibiti RNA kuangalia ikiwa kuna kizuizi. Osha mvua ya RNA na 70% (v / v) ethanoli kuondoa vizuizi.
A-3 kutoweka kwa kutosha kwa viboreshaji vilivyotumiwa kwa kusanikisha kamba ya kwanza ya cDNA
- Amua kuwa joto linalounganisha linafaa kwa viboreshaji vilivyotumika kwenye jaribio. Kwa hexamers isiyo ya kawaida, inashauriwa kuweka joto kwa 25 ° C kwa dakika 10 kabla ya kufikia joto la athari. Kwa utangulizi maalum wa jeni (GSP), jaribu GSP nyingine, au badili kwa oligo (dT) au hexamer ya nasibu.
A-4 Kiasi kidogo cha kuanzia RNA
- - Ongeza kiwango cha RNA. Kwa sampuli za RNA chini ya 50 ng, 0.1 μg hadi 0.5 μg acetyl BSA inaweza kutumika katika muundo wa kwanza wa strand cDNA
A-5 Mlolongo wa lengo haujaonyeshwa kwenye tishu zilizochambuliwa.
—— Jaribu tishu nyingine.
Mmenyuko wa PC-A-6 unashindwa
—— Kwa RT-PCR ya hatua mbili, templeti ya cDNA katika hatua ya PCR haiwezi kuzidi 1/5 ya ujazo wa majibu.
A-1 Kuambatanisha isiyo maalum ya vitangulizi na templeti
—— Mwisho wa 3'mwanzilishi haupaswi kuwa na dG 2-3 au dC. Tumia viboreshaji maalum vya Gene katika usanisi wa kwanza wa strand badala ya viboreshaji vya nasibu au oligo (dT). Tumia joto la juu la kufunika katika mizunguko michache ya kwanza, na kisha joto la chini la kuongeza. Tumia moto-kuanza Taq DNA polymerase kwa PCR kuboresha upekee wa majibu.
Ubunifu duni wa viboreshaji maalum vya jeni
Fuata kanuni zile zile za uundaji wa vifaa vya kukuza.
RNA-3 iliyochafuliwa na DNA ya jeni
Tibu RNA na DNase ya kiwango cha PCR I. Weka athari ya kudhibiti bila unukuzi wa nyuma kugundua uchafuzi wa DNA.
Uundaji wa A-4 ya kipima cha kwanza
-Bunifu la vichangishi bila mlolongo wa nyongeza katika mwisho wa 3.
A-5 Mguu mno2+ mkusanyiko
——Jiongezee Mg2+ mkusanyiko wa kila template na mchanganyiko wa kwanza
A-6 Imechafuliwa na DNA ya kigeni
Tumia vidokezo vinavyoweza kuzuia erosoli na vimeng'enya vya UDG.
A-1 Yaliyomo kwenye bidhaa ya strand ya kwanza ni kubwa mno
- Punguza kiwango cha bidhaa ya strand ya kwanza katika hatua ya kawaida ya athari ya PCR.
A-2 Kiasi cha juu sana cha athari katika athari ya PCR
- - Punguza uingizaji wa mwanzo.
A-3 Mizunguko mingi sana
- Tengeneza hali ya athari za PCR na upunguze nambari ya mzunguko wa PCR.
A-4 Joto la chini sana linalounganisha
- Kuongeza joto linalounganisha ili kuzuia uanzishaji na ugani ambao sio maalum.
A-5 Ukuzaji usio maalum wa vipande vya oligonucleotide vinavyotokana na uharibifu wa DNase wa DNA -Chukua kiwango cha juu cha RNA kuzuia uchafuzi wa DNA.
RT-PCR ni kugeuza nakala ya RNA kuwa cDNA, na kisha utumie cDNA iliyonakiliwa nyuma kama kiolezo cha athari ya PCR kukuza kipande cha lengo. Chagua utangulizi wowote wa nasibu, Oligo dT na viboreshaji maalum vya jeni kulingana na hali maalum za jaribio. Vipimo vyote hapo juu vinaweza kutumika kwa mRNA fupi ya kiini cha eukaryotic bila muundo wa nywele.
Utangulizi bila mpangilio: Inafaa kwa RNA ndefu na muundo wa nywele, na kila aina ya RNA kama rRNA, mRNA, tRNA, nk Zinatumika sana kwa majibu ya RT-PCR ya templeti moja.
Oligo dT: Inafaa kwa RNA na tailing ya PolyA (prokaryotic RNA, eukaryotic Oligo dT rRNA na tRNA haina mikia ya PolyA). Kwa sababu Oligo dT imefungwa kwa mkia wa PolyA, ubora wa sampuli za RNA zinahitajika kuwa juu, na hata idadi ndogo ya uharibifu itapunguza sana kiwango cha usanidi wa cDNA kamili.
Utangulizi maalum wa jeni: Inayosaidia mlolongo wa templeti, inayofaa kwa hali ambazo mlolongo wa lengo unajulikana.
Kuna njia mbili:
1. Njia ya kumbukumbu ya ndani: Kwa nadharia, cDNA ni vipande vya DNA vya urefu tofauti, kwa hivyo matokeo ya electrophoresis ni kupaka. Ikiwa wingi wa RNA ni mdogo, hakuna bidhaa itakayoonyesha katika electrophoresis, lakini hii haimaanishi kuwa hakuna bidhaa itakayokuzwa na PCR. Kwa ujumla, kumbukumbu ya ndani inaweza kutumika kugundua cDNA. Ikiwa kumbukumbu ya ndani ina matokeo, ubora wa cDNA inaweza kuhakikishiwa kimsingi (katika hali chache, ikiwa kipande cha jeni lengwa ni refu sana, kunaweza kuwa na tofauti).
2. Ikiwa kuna jeni inayojulikana iliyopanuliwa na templeti hii, inaweza kudhibitishwa na watangulizi wa jeni hili. Ukuzaji wa kumbukumbu ya ndani haimaanishi kuwa hakuna shida na cDNA. Kwa sababu kumbukumbu ya ndani ina kiwango kikubwa katika cDNA, ni rahisi kuiongezea. Ikiwa cDNA imeharibiwa kwa sehemu kwa sababu anuwai, kutoka kwa mtazamo wa uwezekano, matokeo ya PCR ya jeni la chini la kiwango cha chini yataathiriwa sana. Wakati kumbukumbu ya ndani bado iko juu kwa wingi, ukuzaji hautaathiriwa.
Uharibifu wa sehemu ya RNA. Gundua uadilifu na utakase RNA
Yaliyomo ya RNA ya spishi tofauti inaweza kuwa tofauti, lakini kwa jumla, jumla ya RNA inapaswa kuwa na bendi mbili wazi za 28S na 18S katika gel electrophoresis, na mwangaza wa bendi ya zamani inapaswa kuwa juu mara mbili kuliko ile ya mwisho. Bendi ya 5S inaonyesha kuwa RNA imepungua, na mwangaza wake ni sawa na kiwango cha uharibifu. Kukuza kwa mafanikio kwa kumbukumbu ya ndani haimaanishi kuwa hakuna shida na RNA, kwa sababu rejea ya ndani iko katika kiwango cha juu, RNA inaweza kukuzwa ilimradi uharibifu sio mkali. OD260/ OD280uwiano wa RNA safi iliyopimwa na spectrophotometer inapaswa kuwa kati ya 1.9 na 2.1. Kiasi kidogo cha uchafu wa protini katika RNA itapunguza uwiano. Ilimradi thamani sio chini sana, RT haitaathiriwa. Kilicho muhimu zaidi kwa RT ni uadilifu wa RNA.
Ugani wa jeni ya kumbukumbu ya ndani inaweza kuonyesha tu kwamba RT imefaulu, lakini sio lazima inahusiana na ubora wa strand ya cDNA. Kwa sababu vipande vya kumbukumbu vya ndani kwa ujumla ni ndogo kwa saizi na ya juu kwa kujieleza, ni rahisi kufanikiwa katika nakala ya nyuma. Walakini, saizi na usemi wa jeni lengwa hutofautiana kutoka jeni hadi jeni. Ubora wa cDNA hauwezi kuhukumiwa tu na rejeleo la ndani haswa kwa vipande vya shabaha zaidi ya 2 kb.
Sampuli zingine zina miundo tata ya sekondari, au zina yaliyomo tajiri ya GC, au zina thamani na wingi mdogo. Katika kesi hizi, transcriptase inayofaa ya nyuma inapaswa kuchaguliwa kulingana na saizi ya kipande cha lengo na sampuli. Kwa templeti za RNA zilizo na yaliyomo juu ya GC na muundo tata wa sekondari, ni ngumu kufungua muundo wa sekondari kwa joto la chini, au kwa transcriptase ya kawaida ya nyuma. Kwa templeti hizi, Quant Reverse Transcriptase inaweza kuchaguliwa, kwa kuwa utendaji wake wa kunakili uliobadilishwa ni bora kuliko ile ya M-MLV mfululizo reverse transcriptase, ambayo inaweza kurudisha nakala za templeti anuwai za RNA vizuri na kunakili RNA kwenye strand ya kwanza ya cDNA kwa kiwango cha juu. Unapotumia vifaa vya jumla vya transcriptase ya nyuma, mfumo wa 20 μl unaweza kubadilisha kabisa 1 μg ya jumla ya RNA. Tafadhali zingatia kiwango cha juu cha RT cha kit. Ikiwa templeti imeongezwa kwa ziada, nakala ya nyuma itapendelea RNA na wingi wa juu. Kwa hivyo, ni bora kutozidi uwezo wa kiwango cha juu cha mfumo.
A-1 Tambua ikiwa RNA imeharibiwa sana na ikiwa RT imefanikiwa
Kwa ujumla, sababu ya kutofautishwa kwa ukuzaji wa kumbukumbu ya ndani mara nyingi husababishwa na uharibifu mkubwa wa RNA. Sababu nyingine inayowezekana ni kutofaulu kwa unukuzi. Rejeleo la ndani haliwezi kutumiwa kama kiwango cha kuhukumu ubora wa kamba moja ya cDNA, lakini inaweza kutumika kama kiwango cha kuhukumu ikiwa unukuzi wa nyuma umefanikiwa ikiwa hakuna shida ya ubora wa RNA. Jambo muhimu zaidi katika mchakato wa kunakili nyuma ni kudumisha hali ya joto ya mara kwa mara na mfumo wa athari ya mara kwa mara ili kuboresha ufanisi wa athari.
A-2 Tambua ikiwa viboreshaji vya kukuza jeni za kumbukumbu za ndani ni vya kuaminika na ikiwa kuna shida yoyote na vitendanishi vilivyotumiwa katika PCR.
Kwa hesabu ya jamaa, RNA lazima ihesabiwe kabla ya nakala ya nyuma, ambayo pia inahitajika katika vifaa vingi vya kurudisha nakala, kwa mfano, pima pembejeo ya RNA kama 1 μg. Kwa kuwa cDNA iliyobadilishwa nyuma ni suluhisho mchanganyiko, pamoja na RNA, oligo dT, enzyme, dNTP, na hata mabaki kidogo ya DNA, kupotoka kutasababishwa, kwa hivyo haiwezekani kupima kwa usahihi cDNA. Kwa hivyo, upimaji wa RNA ni muhimu. Kuzingatia ufanisi wa unukuzi wa nyuma ni sawa kati ya sampuli tofauti, kiwango cha cDNA kilichopatikana kinapaswa kuwa sawa, na uchambuzi wa upimaji unaweza kuonyesha kulinganisha kwa viwango vya kujieleza vya jeni tofauti kwa kiwango sawa cha jumla ya RNA. Wakati wa kufanya PCR ya kiwango cha fluorescence ya upimaji, cDNA ya upimaji inaweza kuhitajika baada ya nakala ya kugeuza kwa sababu jeni la kumbukumbu la ndani linaweza kufanywa kama kumbukumbu.
Inahusiana haswa na jeni, na kurudisha nakala ya kipande kirefu haiwezekani kwa jeni nyingi. Kwanza, ufanisi wa unukuzi wa nyuma ni wa chini sana kuliko ule wa PCR. Pili, mkoa tajiri wa GC na muundo wa sekondari wa jeni nyingi huzuia unasaji wa nyuma na PCR. Mwishowe, uaminifu na ufanisi wa ukuzaji wa PCR ni ngumu kuhakikisha wakati huo huo. Katika mchakato wa kunakili nyuma, hakuna mtu anayeweza kuhakikisha kupata kipande kirefu cha jeni za nakala za chini, haswa kwa kutumia oligo dT. Kwa 5 'UTR na GC zaidi, ni ngumu zaidi. Kwa hivyo, bado ni njia inayofaa ya kurudisha nakala na viboreshaji visivyo na mpangilio, pata tovuti za asili za ujasusi kwenye kipande cha shabaha, tukuze kwa sehemu, halafu fanya uzuiaji wa kizuizi na ligation. Kwa ujumla, ni ngumu kukuza moja kwa moja vipande vikubwa zaidi ya 2 kb, lakini sio kila wakati haiwezekani kupata: 1. Kwanza kabisa, hakikisha uadilifu wa RNA / mRNA, na uchimbaji wa TRIZOL unapendelea. 2.M-MLV RT-PCR kit inaweza kutumika moja kwa moja. Panua muda wa kuongeza na kuongeza idadi ya mzunguko katika mchakato wa kukuza vizuri. Vinginevyo, PCR iliyohifadhiwa inaweza kutumika, au kutekeleza athari moja au mbili kwanza na upatanisho unaofaa na muda wa nyongeza kabla ya ukuaji wa kawaida wa PCR, ambayo inaweza kusaidia kupanua vipande. Makini na uaminifu wa polymerase. 3. Long Taq inaweza kutumika katika PCR kupata matokeo bora. 4. Kwa matumizi ya usemi wa protini, uaminifu mkubwa wa polymerase inapaswa kutumika.
Kuna aina mbili za nakala ya nyuma inayotolewa na TIANGEN: Quant / King RTase na TIANScript M-MLV. Tofauti kuu kati yao ni kiasi cha pembejeo cha templeti. Quant ni nakala ya kipekee ya nyuma, ambayo ni tofauti na M-MLV inayotumiwa sana inayotokana na virusi vya leukemia ya Moloney murine. Quant ni nakala mpya ya ufanisi wa hali ya juu iliyoonyeshwa tena na uhandisi Escherichia coli. Kiasi kinafaa kwa kuongeza 50 ng-2 μg ya RNA na shughuli kubwa ya nakala ya nyuma na mavuno mengi. Ikilinganishwa na MMLV ya kawaida au AMV, sifa kubwa ya Quant ni kwamba ina uhusiano mkubwa sana na templeti za RNA na inaweza kubadilisha templeti tata ngumu bila joto la juu. Kwa templeti zilizo na yaliyomo juu ya GC, ufanisi wa nyuma ni wa juu zaidi. Walakini, nakala hii ya nyuma ina shughuli ya RNase H, ambayo inaweza kuathiri urefu wa bidhaa ya cDNA (inayofaa kwa templeti za <4.5 kb). Kwa nakala ya kawaida ya kurudisha nyuma, TIANScript MMLV reverse transcriptase inapendekezwa. RTase hii ni enzyme iliyobadilishwa na shughuli dhaifu sana ya RNase H, ambayo inafaa kwa usanisi wa muda mrefu wa (> 5 kb) cDNA.
Unukuzi wa kurudi nyuma wa hatua moja na ukuzaji wa PCR umekamilika kwenye bomba moja bila kufungua kifuniko cha bomba kati ya usanisi wa cDNA na ukuzaji, ambayo inasaidia kupunguza uchafuzi. Kwa kuwa sampuli zote za cDNA zilizopatikana hutumiwa kukuza, unyeti ni mkubwa, na kiwango cha chini cha 0.01 pg ya jumla ya RNA. Kwa RTPCR ya hatua moja iliyofanikiwa, viboreshaji maalum vya jeni hutumiwa kwa ujumla kuanzisha usanisi wa cDNA. Njia ya hatua mbili, ambayo ni kurudisha nyuma kunakili na ukuzaji wa PCR hufanywa kwa hatua mbili. Kwanza nakala ya nyuma hufanywa kutoka kwa templeti ya RNA kupata cDNA, na cDNA iliyopatikana inakabiliwa na athari moja au zaidi tofauti za PCR. Njia ya hatua mbili inaweza kutumia oligo (dT) au vichocheo visivyo vya kawaida kuongoza usanisi wa strand ya kwanza ya cDNA, na inaweza kubadilisha maelezo yote ya mRNA kutoka kwa sampuli maalum.
Makundi ya bidhaa
KWANINI TUCHAGUE
Tangu kuanzishwa kwake, kiwanda chetu kimekuwa kikitengeneza bidhaa za darasa la kwanza na kufuata kanuni hiyo
ya ubora kwanza. Bidhaa zetu zimepata sifa bora katika tasnia na kudhaminiwa kwa thamani kati ya wateja wapya na wa zamani ..








